2013年,科学家首次证明CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)技术可以在哺乳动物细胞内实现高效的基因组编辑。自此之后,该技术快速发展,为生物医学基础研究和临床应用提供了新模式。
对一个基因编辑方案的技术评估包括编辑效率、编辑安全性(即脱靶效应)和递送效率三个方面。但在以临床应用为目标的应用场景中,其中的编辑安全性则通常被第一优先考虑。就在今年的8月和11月,FDA先后暂停了Beam Therapeutics的CAR-T体外基因编辑治疗IND申请和Verve Therapeutics的PCSK9体内基因编辑治疗管线的一期临床试验,要求新药研发机构提供更多的数据,评估在利用基因编辑技术展开治疗的过程中相关的脱靶编辑风险,以及由此带来的染色体易位风险。
由于CRISPR系统巨大的临床应用潜力,对于基因编辑过程中脱靶效应进行评估的探索,几乎与CRISPR系统被证实可以应用于编辑哺乳动物基因组同时出现。至今为止,科学家们已经开发了诸多方法来评估基因编辑过程中伴随发生的潜在脱靶编辑事件。这些方法可以分析不同基因编辑场景下的基因组DNA,一般被归纳为细胞外(in vitro,如Circle-seq)、体外培养细胞中(in cellula,如GUIDE-seq)、体内组织细胞中(in vivo,如Discover-seq)三类。这些方法的共同思路,是在基因编辑过程中,将所有被编辑位点(包括靶点和脱靶位点)打上“标签”,再通过对带标签基因组序列的富集来检测基因组的哪些区域发生了CRISPR相关的基因编辑。
作为目前被较广泛使用的方法,GUIDE-seq通过在基因编辑的过程中给细胞内加入大量外源dsODN(double-stranded oligodioxynucleotide)来完成加标签的过程。但是,由于外源dsODN对细胞具有毒性,使得GUIDE-seq无法被应用于体内基因编辑的脱靶检测。另一种被报道可以用于体内检测的Discover-seq技术,则通过对DNA修复蛋白MRE11的免疫共沉淀,来鉴定脱靶编辑发生的基因组位置。因此,其能够找到的脱靶编辑位点仅限于某一个时间点上细胞内正在修复中的DNA双链断裂处的瞬时快照,而不能够反应整个编辑时间窗口中发生的所有编辑事件。
西湖大学生命科学学院马丽佳研究员和团队成员们也一直在寻找新方法,希望克服以上缺点,优化基因编辑脱靶位点的检测。
“PEAC-seq adopts Prime Editor to detect CRISPR off-target and DNA translocation”于2022年12月12日在线发表在Nature Communications。
论文链接:https://www.nature.com/articles/s41467-022-35086-8
原文截图
Prime Editor(PE)是David Liu博士研究小组于2019年发表的一种新型基因编辑工具,其本身的应用场景是基因编辑而非鉴定脱靶位点。PE系统将Cas9 nickase(Cas9n)和逆转录酶MMLV融合,并创新性地将想编辑成的DNA信息添加在gRNA的3’端(pegRNA)作为逆转录酶的反转录模板,通过反转录,目标DNA序列会被引入基因组。在随后的几年里,通过David Liu、Caixia Gao等实验室的一系列优化工作,PE系统目前已经能够达到非常高的编辑效率。
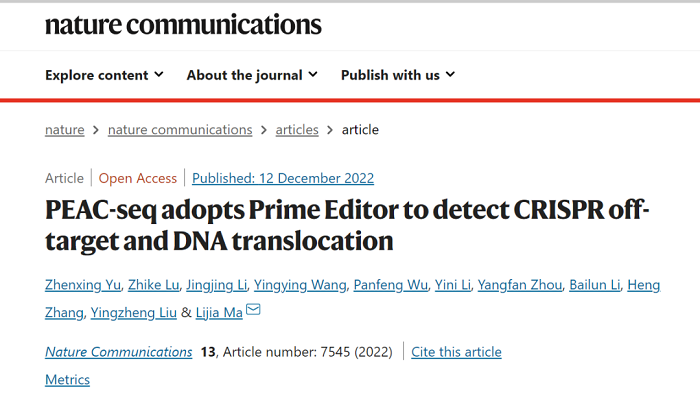
受此启发,马丽佳实验室的研究人员对PE系统进行了改造。首先,他们将pegRNA上的反转录模板改造为一段优化过的固定插入序列(tag)。在PE系统的作用下,发生了编辑的基因组位点都会带有这段标签序列。随后,他们改用野生型Cas9而非Cas9n与MMLV融合,使得检测到的脱靶编辑都来自于Cas9引起的双链DNA断裂。最后,在Tn5转座酶的配合下,利用两对富集引物,所有带有tag的基因组位置通过PCR反应被富集,并最终从高通量测序数据中被鉴定出来。整套方案被命名为PEAC-seq(图1),为Primer Editor Assisted off-target Characterization by sequencing的缩写。
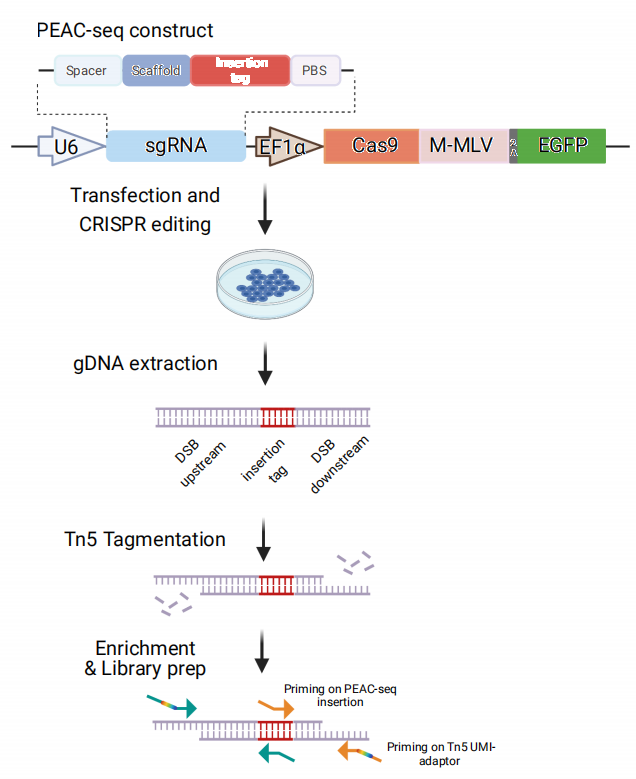
图1 PEAC-seq流程图
研究人员在HEK293T细胞中选择了六个被广泛用于测试脱靶编辑的位点,将PEAC-seq鉴定到的脱靶位点与GUIDE-seq的检测结果进行比较,并通过Amplicon-seq进行验证,发现PEAC-seq与GUIDE-seq的结果具有很好的一致性。
在对PEAC-seq的测序信号进行仔细查验的过程中,研究人员意外地发现在某些脱靶位点,有部分信号不是来自于该位点本身随PE系统被插入的PEAC-seq tag。由于tag的插入具有方向性,两对用于富集tag的PCR引物各自只能扩增出tag上游、或者是tag下游的基因组序列。随着分析的深入,研究人员推测异常信号很可能来自于基因组中多个被编辑位点在各自发生DNA双链断裂后,由于在三维空间上靠近,而产生易位连接在一起,并提出了在PEAC-seq系统中,几种可能发生的DNA易位连接模式(图2)。

图2 PEAC-seq检测DNA translocation的五种可能情况(底部粉色图形表示异常信号)
经过验证,研究人员证实了他们的猜测,发现染色体易位情况在很多靶位点(on-target)和脱靶位点(off-target)上都存在(图3)。但染色体易位的发生概率与位点本身的编辑效率并没有明显正相关,提示了有其他因素的影响。相比于脱靶效应,染色体易位带来的基因组毒性会影响更大范围的基因表达,造成细胞功能的异常。然而,以往的基因编辑脱靶检测方法,需要通过已知的基因组上下游序列、逐个位点去鉴定染色体易位,无法对染色体易位情况进行全基因组评估。PEAC-seq弥补了这方面的不足,为系统性鉴定由基因编辑引起的染色体易位提供了解决方案,也为更为安全地实施基因编辑提供了参考依据。

图3 Identification and validation of DNA translocations
最后,研究人员将体外转录的Cas9-MMLV的mRNA和pegRNA通过显微注射的方式,递送入小鼠受精卵中,一段时间后收集小鼠胚胎检测脱靶编辑情况。这个实验验证了PEAC-seq在体内基因编辑场景中也可以被安全地用于检测脱靶位点(图4)。
图4 PEAC-seq in vivo application
研究人员还对基因组中脱靶编辑和染色体易位发生的规律展开了进一步分析。他们发现,发生脱靶编辑的位点,与被基因编辑的细胞系中代表染色体开放区域((ATAC-seq)、基因的转录调节区域(H3K4me3,H3K9ac,and H3K27ac,POLR2A,EP300,H2AFZ)的信号具有明显的共富集。而染色体易位,则与细胞本身的双链断裂热点具有共富集。
总之,PEAC-seq提供了一种新型的Cas9基因编辑脱靶位点检测方案,在体外培养细胞和体内受精卵中都被证明可以安全、准确地鉴定出脱靶位点。更重要的是,PEAC-seq为基因编辑引起的染色体易位鉴定提供了解决方案。随着基因编辑技术被应用在越来越多的基因治疗临床试验中,之前被忽视的染色体易位现象需要在灵敏的检测方法所产生的数据支持下,被更为深入地分析。