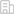剪接体是一个巨大且高度动态的分子机器,其主要组成元件为5种snRNP(分别为U1,U2,U4,U5和U6)。其中U2 snRNP在识别内含子和剪接体的早期组装中起至关重要的作用。人源U2 snRNP的组装十分复杂,对于其组装过程中的调控因子知之甚少。目前,发现三种RNA解旋酶与U2 snRNP的组装相关——DDX42,DDX46和DHX15,但是它们的作用机制尚不明确。
解决这些问题的关键是阐明RNA解旋酶的结构和功能。SF3b是U2 snRNP的核心组分,本研究报道了DDX42-SF3b复合物和DDX42-U2复合物的高分辨率电镜结构(图1),DDX42通过N端序列结合在SF3B1上,其N-plug占据SF3B1的RNA通道。这和DDX46与SF3B1的结合方式十分相似。在DDX42-U2复合物中,DDX42的N端仍然锚定在SF3B1上,但C端解旋酶结构域结合的位置已被U2 snRNA和剪接因子TAT-SF1取代。
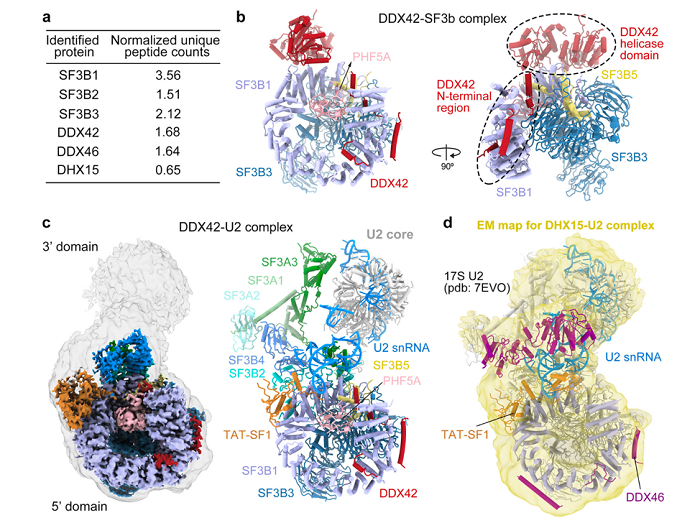
图1 DDX42-SF3b和DDX42-U2复合物的电镜结构
通过与先前报道的17S U2[1]以及剪接体Bact[2]复合物的结构比较(图2),研究者发现SF3B1的RNA通道依次被三个结构元件所占据:DDX42的N-plug,DDX46的acidic loop以及pre-mRNA的多聚嘧啶区(polypyrimidine-tract, PPT),因此,研究者推测:它们与RNA通道之间的结合应该是互斥的,之后,研究者通过体外生化实验证实了这个猜想。由此可见,DDX42从SF3B1上释放是招募DDX46、形成成熟的17S U2 snRNP的先决条件,而DDX46的解离则是SF3B1结合pre-mRNA、剪接体组装的必要条件。
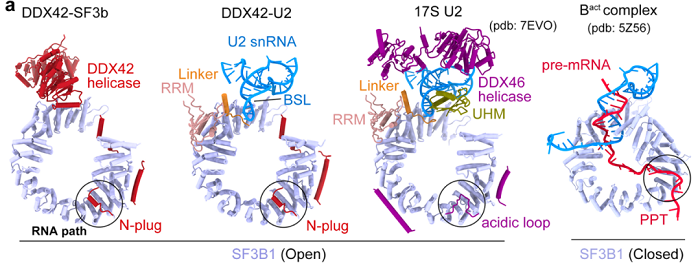
图2 不同结构中SF3B1的RNA通道的比较
SF3B1的突变会导致RNA剪接异常,并与多种癌症的发生密切相关。研究者发现这些突变位点大多数都位于SF3B1与DDX42或DDX46的相互作用界面上,体外实验也证实这些突变会削弱与DDX42和DDX46的结合。由此,研究者推测SF3B1的突变可能通过影响U2 snRNP的组装或剪接反应的保真性从而导致RNA剪接异常。
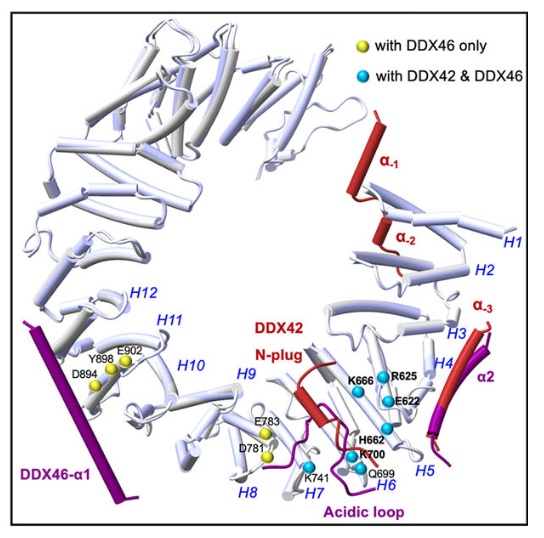
图3 SF3B1癌症相关突变
RNA解旋酶是驱动剪接体组分和构象重排的关键因子,本研究通过结构与生化分析,揭示了RNA解旋酶DDX42和DDX46在U2 snRNP组装过程中的不同作用机制,也为与癌症相关的SF3B1突变的致病机理提供了新的见解。
西湖大学生命科学学院施一公教授、原西湖大学助理研究员张晓峰为本文的共同通讯作者。西湖大学博士生杨丰华、卞彤和副研究员占谢超为本文的共同第一作者。新疆医科大学陈哲、西湖大学博士生邢芷涵、Foghorn Therapeutics公司Nicolas A. Larsen参与了本研究的部分工作。电镜数据采集于西湖大学冷冻电镜平台,计算工作得到西湖大学高性能计算平台的支持,质谱分析得到西湖大学质谱与代谢组学平台的支持。本研究获得了国家自然科学基金委、西湖教育基金会、西湖大学、中国博士后科学基金以及博士后创新人才支持计划的相关经费支持。