近日,我校计算机学院彭玺教授课题组与四川大学华西医院罗晗研究员课题组、生物治疗国家重点实验室陈路研究员课题组合作在基于细胞量化的元细胞推断方法上取得新进展,相关成果近日被Nature Communications正式接收。计算机学院(软件学院、智能科学与技术学院)为该论文第一完成单位,彭玺教授为唯一通讯作者,2020级直博研究生李云帆为唯一第一作者。
论文:MetaQ: fast, scalable and accurate metacell inference via single-cell quantization[1]
【背景】:近年来,单细胞测序技术取得了快速的进展,能够捕获的细胞数量不断增加,其在揭示细胞异质性和重建细胞发育轨迹方面展现出巨大的优势。然而,随着数据规模的增长,单细胞测序数据的分析也面临严峻的计算挑战。例如,典型的单细胞数据分析流程(包括数据整合、聚类、可视化和差异表达分析)在处理五十万个细胞时需耗时约16小时,而当细胞数量增加至六十万时,即便在配备512 GB内存的专业计算平台上,也可能因内存不足而导致程序崩溃。
为应对大规模单细胞测序数据带来的计算开销,研究人员提出了多种高效的单细胞分析工具,主要用于数据插补、整合、聚类和细胞类型注释等任务。然而,这些工具通常专门针对特定任务设计,难以直接集成到现有的单细胞数据分析框架中。为实现更通用且高效的单细胞数据处理,一种解决方案是对原始数据进行压缩,进而降低数据冗余,赋能传统分析工具以更高效地处理大规模测序数据。针对单细胞数据压缩,一种代表性方法是元细胞(Metacell)推断,其通过聚合生物学上相似的细胞群体,将若干个单细胞压缩为单个代表性的元细胞,从而在最大程度上保留生物信息的情况下有效减少了细胞数量。
元细胞推断方法在大规模数据处理中具有显著优势。一方面,元细胞带来的数据压缩减少了测序数据分析的计算开销;其次,通过聚合特征相似的细胞,元细胞缓解了数据的稀疏性,在一定程度上提升了下游分析(如细胞类型注释、发育轨迹推断等)的鲁棒性。然而,尽管元细胞推断方法在一些应用场景下取得了令人满意的效果,其在大规模数据集上的准确性和效率仍存在不足。例如,当前最优的SEACell算法[2]通过构建全局的邻接矩阵进行单细胞聚类,并根据聚类结果来推断元细胞。该算法在处理较小规模数据时取得了良好的效果,但当处理10万个单细胞时则需要大于一天的时间开销,且由于其指数级的内存开销,难以处理更大规模的单细胞数据。换而言之,现有的元细胞推断方法本质上将计算瓶颈从下游分析转移到了元细胞推理阶段,而未真正解决计算复杂度问题。
【创新】:本文提出了首个可处理任意规模单细胞数据的元细胞推断算法MetaQ,将计算复杂度从现有方法的指数级降低到线性。受多细胞生物细胞分化过程的启发,MetaQ将每个元细胞视为一个特化细胞亚群的公共祖先,其能够有效地派生出该亚群的全部细胞。基于该思想,MetaQ将所有细胞量化到一个具有若干可学习条目的码本,码本的每个条目用于重构其所量化的原始细胞。为了更好的重构效果,相似的细胞将被量化到同一码本条目中。换而言之,该生成式细胞量化过程有助于识别同质细胞子集,每个码本条目本质上对应一个元细胞表征,从而实现准确的元细胞推断。此外,不同于现有的元细胞方法仅面向单组学测序数据,本文提出的MetaQ方法支持多组学配对数据的元细胞推断,大大提升了方法的适用范围。

图1 MetaQ算法示意图
【方法】:本文提出的基于细胞量化的元细胞推断方法整体框架如图1所示:(a)对于给定的单组学或多组学单细胞数据,首先使用编码器学习细胞表征;(b)在表征空间,引入一个由若干可学习条目组成的元细胞码本,每个条目对应一个元细胞的表征。之后,通过将每个细胞划分到最相似的码本条目来实现细胞量化。为了防止码本坍缩,记录不同条目的使用情况,并调整被过多或过少使用的码本条目;(c)每个码本条目通过解码器重构其所量化的全部细胞。为了更好的重构效果,更相似的细胞将被划分到同一码本条目中;(d)训练完成后,对每个码本条目对应的原始细胞数据进行平均来推断元细胞;(e)推断出的元细胞作为原始数据的代理,可以无缝地用于各类单细胞下游分析。
【结果】:本文在六个数据集上验证了所提出MetaQ方法的性能和效率。与当前最优的SEACell算法[2]相比,MetaQ在处理10万个细胞时节约了约100倍的时间和25倍的内存开销。实验表明MetaQ推断的元细胞在各种下游任务上均优于现有的方法,包括细胞类型注释、发育轨迹推断、批次整合、聚类和差异表达分析等。以下展示部分实验结果,更多结果和分析详见原文。
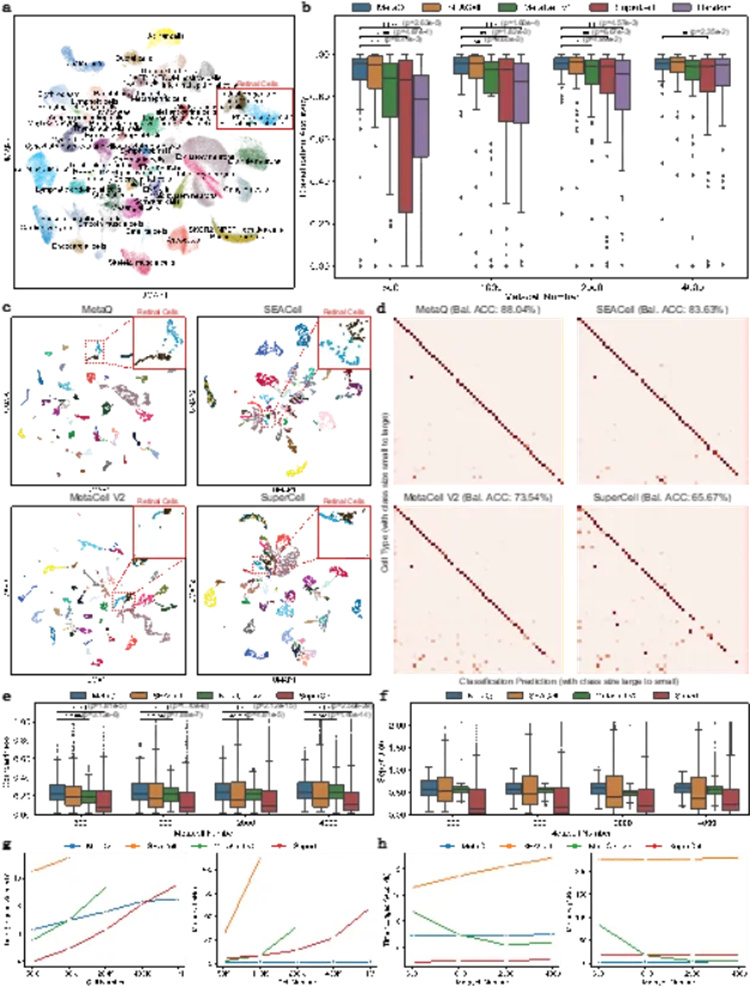
图2 MetaQ在Human Fetal Atlas数据集的元细胞推断结果
【总结】:本文提出了一种高效且准确的元细胞推断算法,将现有方法的复杂度从指数级降低至线性。所推断的元细胞作为原始细胞的压缩表示,在保留生物特性的同时显著减少细胞数量,从而使经典单细胞分析工具处理数百万个细胞变为可能。考虑到高通量单细胞测序技术的进步和数据的规模不断增加,MetaQ有望成为一个具有广泛应用场景的数据处理工具。本研究是机器学习算法在生物信息分析中的一次成功应用,也是彭玺教授研究组近年来对AI4Science研究的又一次有益尝试。